南湖新闻网讯(通讯员 高鹏飞)10月11日,我校植物科学技术学院王旭彤教授课题组在国际学术期刊Briefings in Bioinformatics在线发表题为最新研究成果。该研究基于卷积神经网络融合CA注意力机制开发出了作物表型预测模型SoyDNGP,并将其应用于大豆性状预测中,与DNNGP、DeepGS等经典预测模型相比,其性能具有明显提升。团队基于该模型搭建了大豆表型预测网站,为该领域研究团队提供免费、开放的预测服务(http://xtlab.hzau.edu.cn/SoyDNGP),为便于各团队自主构建、改进模型算法,得到更加适用的预测模型,团队同时开发出便捷高效的PyPI软件包SoyDNGPNext,为基于基因变异的作物表型预测的相关研究提供了新工具、新方法。
SoyDNGP网页界面
大豆作为我国重要的经济作物,长年高度依赖国外进口,严重影响我国粮食、经济安全。因此,加速我国大豆育种进程,提高国产大豆产量及品质,是作物育种领域至关重要的研究方向。该研究旨在通过根据作物基因型,对种质资源表型的进行高效、准确预测,指导大豆选择育种,从而促进大豆育种研究的进程。算法通过将作物基因型映射至三维矩阵,得到了相较于以往利用One-hot向量所构建的特征图信息密度更高、计算速度更快的模型输入,使用主体为十二层卷积,加入注意力机制的模型在以USDA数据库的5000份种质样本作为训练集,2018年郑州559份大豆样本作为验证集的数据集上进行训练、预测。
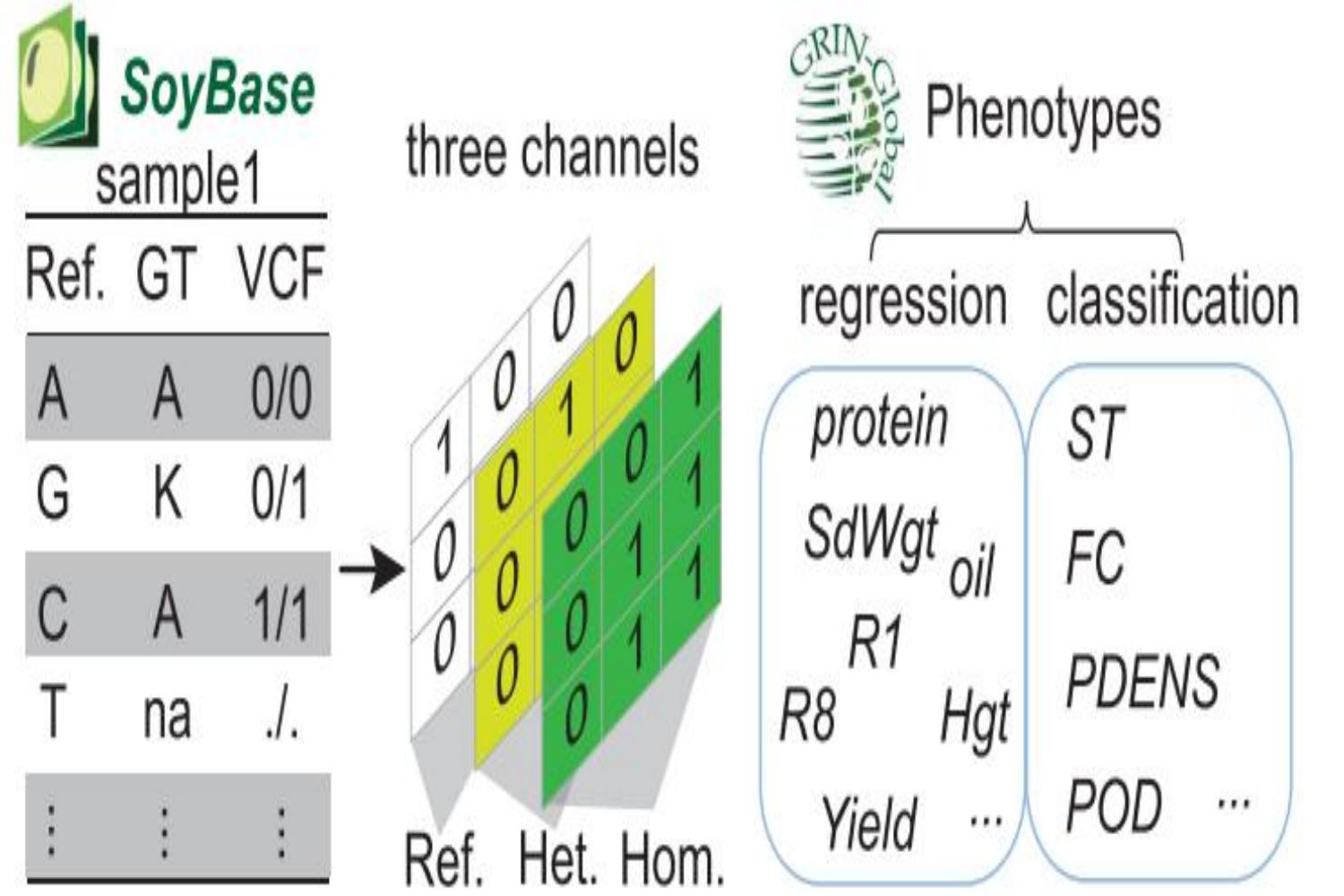
模型输入映射示意图
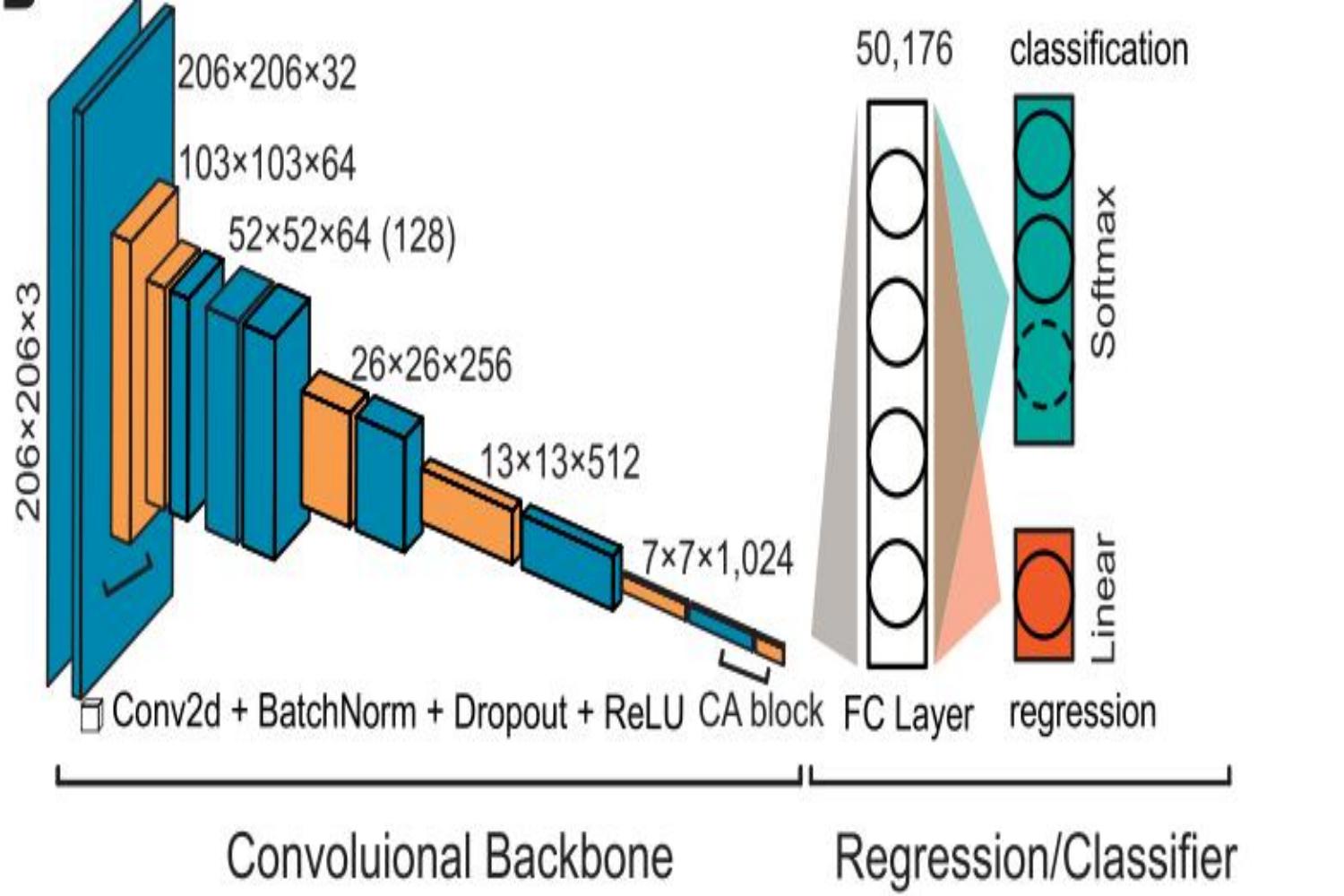
网络结构示意图
在测试集中,该模型在大豆粒重、产量、株高含量等关键性状上相关系数均达到70%,其中粒重达到89%。在验证集上,模型依旧表现出较为良好的性能。模型在野生种与栽培种的蛋白、产量等性状预测比较中,得到了与先验知识相符的实验结果,且在玉米,小麦,番茄以及水稻等作物上均有较为可观的预测效果,证明了该模型具有较好的鲁棒性。
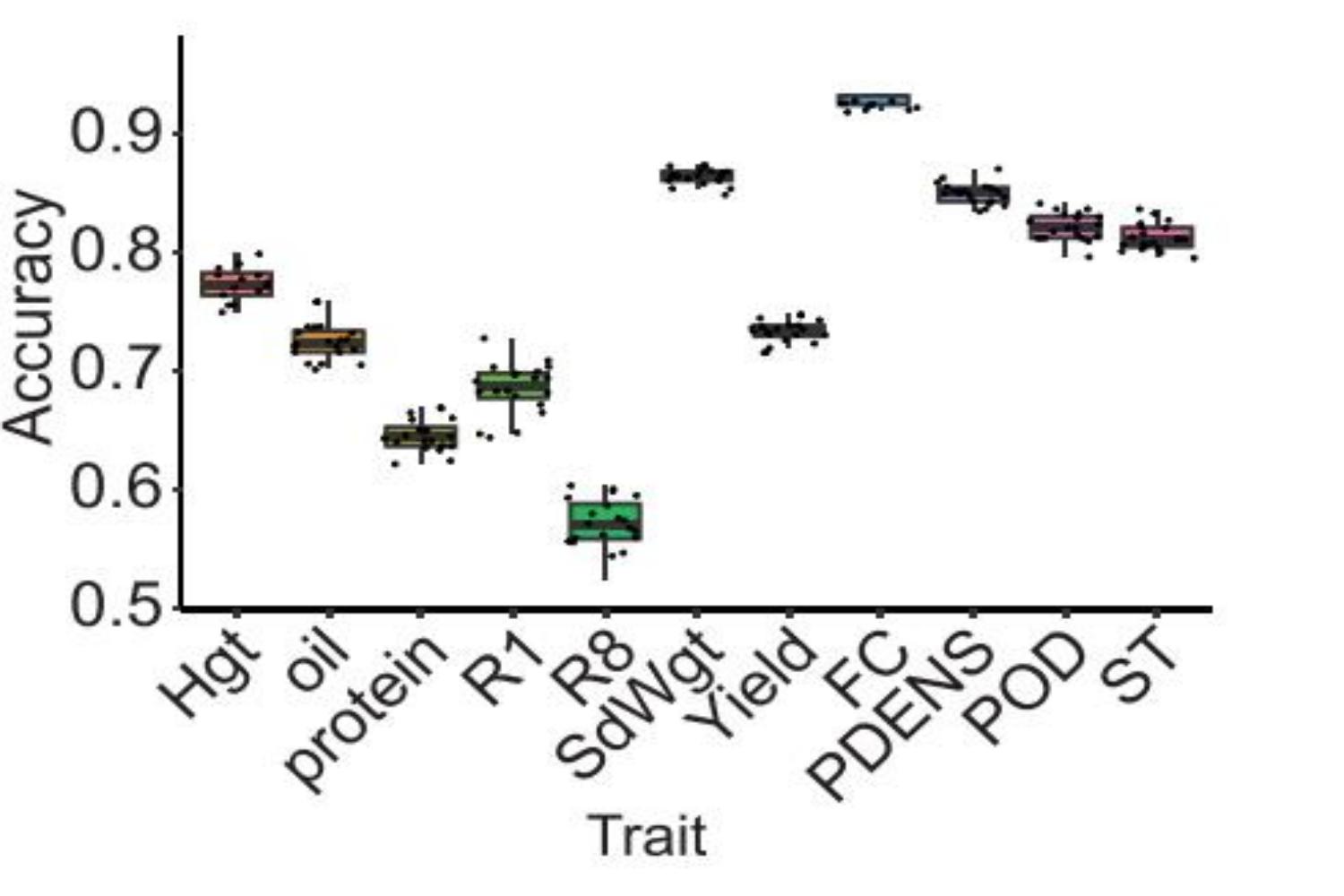
测试集准确率箱型图

验证集相关系数结果图
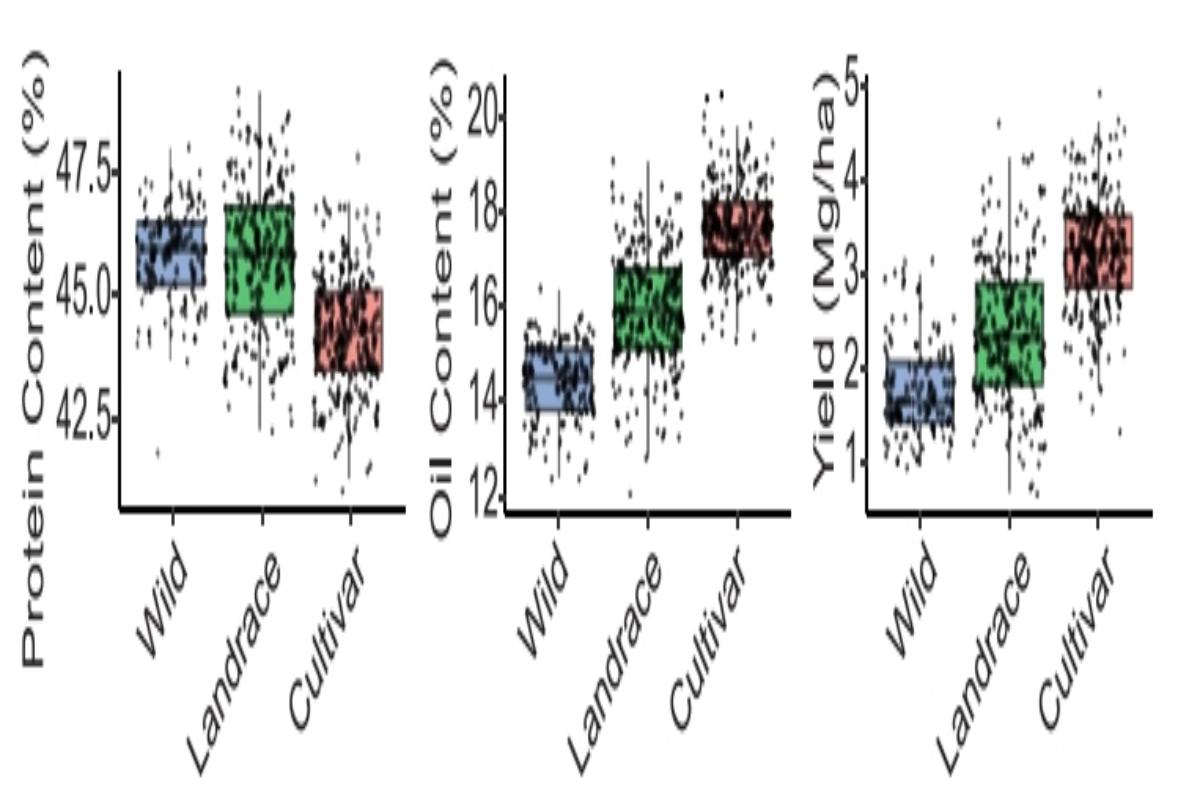
验证集野生型栽培种预测对比
该项目已获得国家重点研发计划《主要农作物多基因高效聚合技术》(2022YFD1201502) 的经费支持,并由王旭彤教授课题组负责《复杂性状全基因组选择新方法开发》子项目,致力于大豆全基因组选择模型的开发和应用。SoyDNGP模型以其出色的全基因组预测能力,在23个大豆性状的选择模型中实现了高准确度的表型预测,为早期种质资源的快速筛选和种质改良提供了强大的技术支持。同时,将模型应用于杂交双亲的配种后代表型预测,能为育种家筛选提供指导,从而提高目标亲本选择的效率和准确性。
据悉,本研究得到了广州大学孔凡江、方超团队的支持,为模型验证提供了宝贵的种质资源及田间表型数据,并得到了项目首席严建兵教授的帮助。该文章由我校植物科学技术学院智慧农业专业本科生高鹏飞、赵浩楠担任共同第一作者,智慧农业专业本科生罗铮、课题组研究生林依繁、冯万杰、李亚玲为共同作者,我校王旭彤教授、李霞教授与方超副教授担任本文共同通讯作者。
【英文摘要】
Soybean is a globally significant crop, playing a vital role in human nutrition and agriculture. Its complex genetic structure and wide trait variation, however, pose challenges for breeders and researchers aiming to optimize its yield and quality. Addressing this biological complexity requires innovative and accurate tools for trait prediction. In response to this challenge, we have developed SoyDNGP, a deep learning-based model that offers significant advancements in the field of soybean trait prediction. Compared to existing methods, such as DeepGS and DNNGP, SoyDNGP boasts a distinct advantage due to its minimal increase in parameter volume and superior predictive accuracy. Through rigorous performance comparison, including prediction accuracy and model complexity, SoyDNGP represents improved performance to its counterparts. Furthermore, it effectively predicted complex traits with remarkable precision, demonstrating robust performance across different sample sizes and trait complexities. We also tested the versatility of SoyDNGP across multiple crop species, including cotton, maize, rice and tomato. Our results showed its consistent and comparable performance, emphasizing SoyDNGP’s potential as a versatile tool for genomic prediction across a broad range of crops. To enhance its accessibility to users without extensive programming experience, we designed a user-friendly web server, available at http://xtlab. hzau.edu.cn/SoyDNGP. The server provides two features: ’Trait Lookup’, offering users the ability to access pre-existing trait predictions for over 500 soybean accessions, and ’Trait Prediction’, allowing for the upload of VCF files for trait estimation. By providing a highperforming, accessible tool for trait prediction, SoyDNGP opens up new possibilities in the quest for optimized soybean breeding.
审核人:王旭彤
